Tìm hiểu cách thuật toán lập trình động được ứng dụng trong tin sinh học để so sánh trình tự DNA, RNA và protein. Phân tích ưu điểm và các công cụ phổ biến sử dụng thuật toán này.
Mục lục bài viết
Lập Trình Động (Dynamic Programming) trong Tin Sinh Học là Gì?
Dynamic programming là một kỹ thuật tối ưu hóa mạnh mẽ trong khoa học máy tính, được ứng dụng rộng rãi trong so sánh trình tự sinh học. Trong tin sinh học, nó giúp giải quyết các bài toán tìm sự tương đồng tối ưu giữa hai hoặc nhiều chuỗi DNA, RNA, hoặc protein – những bài toán mà phương pháp duyệt đơn thuần không thể xử lý hiệu quả.
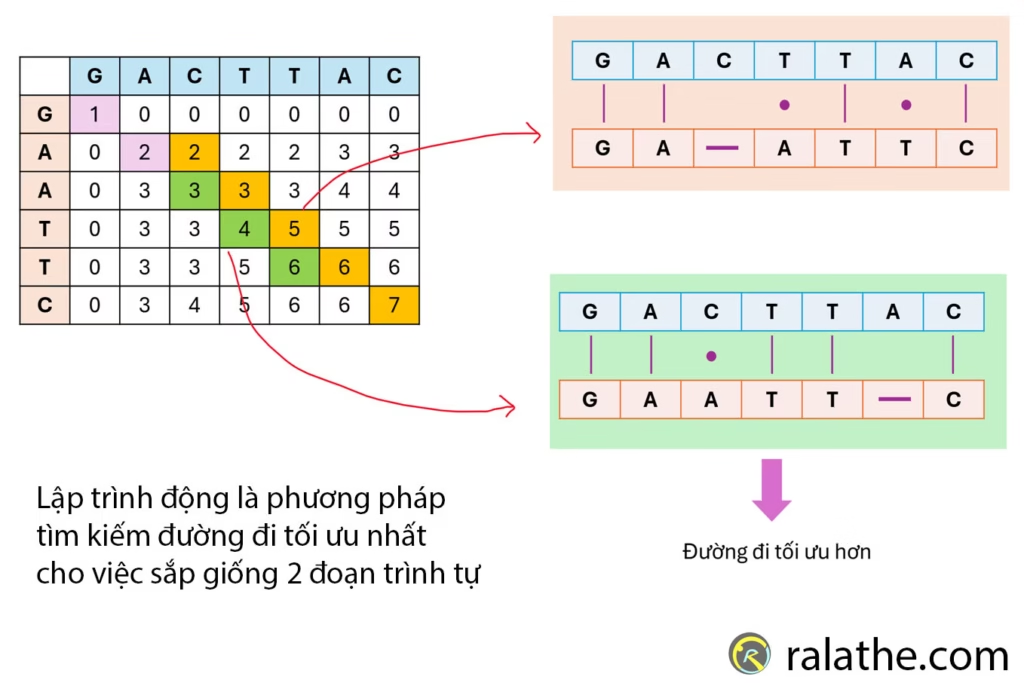
Ứng Dụng của Thuật Toán
Trong bioinformatics, lập trình động đặc biệt hữu ích trong các nhiệm vụ:
- 🔍 Căn chỉnh trình tự toàn cục (global alignment): Dựa trên thuật toán Needleman-Wunsch, giúp căn chỉnh toàn bộ hai chuỗi với nhau, thường dùng khi chúng có độ dài tương đương.
- 🧩 Căn chỉnh cục bộ (local alignment): Dựa trên thuật toán Smith-Waterman, giúp tìm vùng tương đồng tốt nhất giữa hai chuỗi bất kỳ, dù có độ dài khác nhau.
- 🧬 Tìm đường đi tối ưu trong cấu trúc bậc hai RNA
- 🔢 Tính toán khoảng cách chỉnh sửa (edit distance) giữa các chuỗi.
>>>> Tìm hiểu cơ sở dữ liệu Ensembl, một đại diện đến từ châu Âu
Tại Sao Dynamic programming Quan Trọng Trong Tin Sinh Học?
- Đảm bảo kết quả tối ưu: So với các phương pháp heuristic như BLAST, lập trình động cho kết quả chính xác tuyệt đối (exact).
- Linh hoạt với các loại chuỗi khác nhau: Có thể áp dụng với DNA, RNA hoặc protein.
- Tùy chỉnh ma trận điểm và phạt gap: Cho phép điều chỉnh theo mục tiêu nghiên cứu (ví dụ: penalize gaps, mismatches,…).
>>>> BLAST – công cụ mạnh mẽ cho việc phân tích tiến hóa và chức năng
Ưu và Nhược Điểm của lập trình động
Ưu điểm:
- Độ chính xác cao, tối ưu toàn cục hoặc cục bộ.
- Giải được các bài toán phức tạp như RNA folding, sequence alignment, motif search.
Nhược điểm:
- Chi phí tính toán cao (đặc biệt với nhiều chuỗi hoặc chuỗi dài).
- Không phù hợp với việc tìm kiếm nhanh trên cơ sở dữ liệu lớn (so với BLAST).
>>>> Thuật toán ma trận điểm, một thuật toán sắp giống đơn giản
Công cụ giúp bạn trong Tin Sinh Học
- EMBOSS Needle & Water: Giao diện trực quan để chạy thuật toán Needleman-Wunsch và Smith-Waterman.
- Biopython: Thư viện Python hỗ trợ sẵn các hàm alignment bằng dynamic programming.
- ClustalW, MAFFT: Dùng cho nhiều chuỗi (multiple sequence alignment), áp dụng lập trình động mở rộng.
Kết Luận
Lập trình động là nền tảng thuật toán quan trọng trong tin sinh học. Chúng đặc biệt hữu ích trong các nhiệm vụ liên quan đến so sánh trình tự. Dù tốn tài nguyên hơn các phương pháp heuristic, nhưng nó mang lại độ chính xác tối ưu.
Công cụ rất phù hợp cho các nghiên cứu chuyên sâu và phân tích chi tiết về chức năng gen hay vùng bảo tồn tiến hóa. Bất kỳ ai làm việc trong lĩnh vực bioinformatics đều nên nắm vững công cụ này.
